PLAID : Génération multimodale de protéines par diffusion latente
Sources: http://bair.berkeley.edu/blog/2025/04/08/plaid, http://bair.berkeley.edu/blog/2025/04/08/plaid/, BAIR Blog
Aperçu
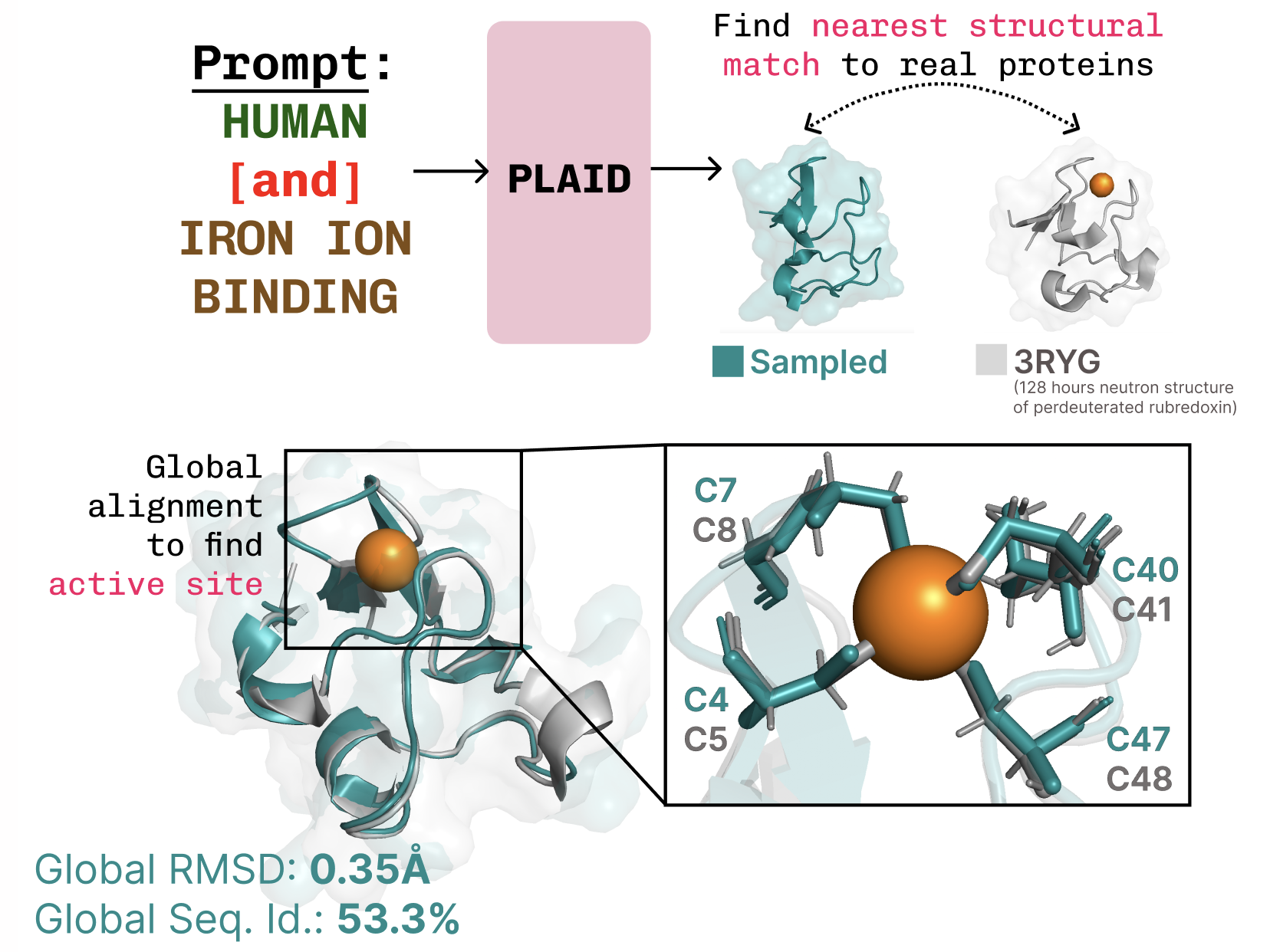
PLAID est un modèle génératif multimodal qui produit simultanément la séquence 1D d’une protéine et sa structure 3D en apprenant l’espace latent des modèles de pliage des protéines. Cette approche s’inscrit dans le contexte récent de l’IA en biologie, suite à la reconnaissance du rôle des modèles de pliage comme AlphaFold2. PLAID apprend à échantillonner l’espace latente d’un modèle de pliage afin de générer de nouvelles protéines et peut être guidé par des invites composées spécifiant la fonction et l’organisme. L’entraînement peut être réalisé uniquement sur des données de séquences, car les bases de données de séquences sont beaucoup plus vastes que celles des structures. L’idée centrale est d’effectuer une diffusion dans l’espace latent d’un modèle de pliage plutôt que directement dans l’espace des séquences ou des structures. Après échantillonnage, l’embedding est décodé en séquence et en structure grâce à des poids gelés d’un modèle de pliage. Dans les faits, PLAID utilise ESMFold — successeur d’AlphaFold2 — qui remplace une étape d’extraction par un modèle de langage protéique, lors de l’inférence. Cette architecture exploite les priors appris par les modèles de pliage pré-entraînés pour les tâches de design protéique, tout en s’appuyant sur les données de séquence pour l’entraînement. Un point clé est la gestion de l’espace latent de grande dimension des modèles basés sur des transformeurs. Pour y remédier, CHEAP (Compressed Hourglass Embedding Adaptations of Proteins) est proposé pour apprendre une compression de l’embedding joint séquence-structure. L’espace latent apparaît comme hautement compressible et une interprétation mécanistique permet d’aboutir à un modèle génératif atomique dans certains cas. Bien que l’étude se concentre sur la génération séquence–structure, elle suggère que la méthode peut être adaptée à une génération multimodale dans d’autres domaines lorsque un préditeur entre les modalities est disponible. PLAID propose aussi une interface de contrôle via des invites de texte, inspirée des commandes textuelles utilisées pour guider la génération d’images. L’objectif recherché est un contrôle total par texte, mais l’interface actuelle permet des contraintes composites selon deux axes: fonction et organisme. Les auteurs démontrent des capacités, par exemple l’apprentissage de motifs de coordination tétraédrique dans les métallo‐protéines (c’est-à-dire cystéine-Fe2+/Fe3+) tout en conservant une diversité au niveau de la séquence. En comparaison avec des baselines basés sur des structures atomiques, PLAID montre une meilleure diversité et capture des motifs beta qui sont plus difficiles à apprendre. Si vous souhaitez collaborer pour étendre la méthode ou tester dans le laboratoire expérimental, les auteurs invitent à les contacter. Ils indiquent également des pré-prints et des bases de code pour PLAID et CHEAP.
Caractéristiques clés
- Génération multimodale: séquences et coordonnées structurales atomiques
- Diffusion latent: échantillonnage dans l’espace latent d’un modèle de pliage, puis décodage
- Entraînement sur des séquences uniquement: exploite de grandes bases de données de séquences
- Décodage avec poids gelés: décode la structure à partir de l’embedding échantillonné via ESMFold
- Compression CHEAP: apprentissage d’un embedding joint compression
- Invites fonction et organisme: contrôle par prompts composés
- Interprétabilité et motifs: analyse des canaux latents et motifs émergents
- Diversité: les échantillons PLAID présentent une diversité meilleure que certains baselines
- Extensibilité: adaptation possible à d’autres modalities lorsque des prévisionnaires existent
- Intégration avec les priors existants: exploite des modèles de pliage pré-entraînés
Cas d’utilisation courants
- Conception de protéines avec des fonctions et organismes ciblés
- Exploration des espaces de design multimodaux via des invites fonctionnelles ou taxonomiques
- Génération de candidats avec des séquences et structures diversifiées respectant les relations fonction-structure apprises
- Extension vers des systèmes plus complexes (par exemple, complexes protéiques ou interactions avec des ligands ou des acides nucléiques)
- Exploitation de grandes bases de données de séquences pour former des modèles capables de générer des structures à partir d’embeddings latents
Mise en place & installation
Non fourni dans la source.
# Mise en place non détaillée dans la source.Démarrage rapide
Note : ceci est une illustration conceptuelle basée sur PLAID; les API exactes et l’environnement d’exécution dépendent du code publié.
# PLAID quick-start (illustratif)
# Flux prévu mais conceptuel, pas un code prêt à l’emploi
# 1) Échantillonner un embedding latent à partir du modèle de diffusion
z = sample_latent_embedding()
# 2) Décoder la séquence et la structure en utilisant les poids gelés de ESMFold
sequence = decode_sequence_from_embedding(z)
structure = decode_structure_from_embedding(z, frozen_weights="ESMFold")
print(sequence)
print(structure)Avantages et limites
- Avantages
- Génération conjointe de séquences et structures guidée par des prompts
- Exploite de grandes bases de données de séquences pour l’entraînement
- Décodage de structure via des poids gelés, simplifiant l’intégration du pliage
- CHEAP aborde la compression et la gestion d’un embedding joint
- Montre un contrôle par prompts et promet un contrôle textuel complet futur
- Diversité et recapture des motifs superent certains baselines
- Limites
- L’espace latent des transformeurs peut être énorme et nécessite une régularisation
- Le succès dépend de la qualité des priors et de l’alignement séquences–structures
- L’application pratique dépend de prompts variés et d’un décodage robuste pour des structures complexes
- L’extension multimodale reste en développement et dépend des progrès des prévisionnaires entre modalités
Alternatives (brève comparaison)
| Approche | Modalité | Points forts | Limitations |---|---|---|---| | PLAID | Séquence + structure | Génération multimodale via diffusion latente; entraînement sur séquences | Dépend d’un modèle de pliage comme backbone; la qualité dépend de la régularisation latente |Générateurs traditionnels de structure | Structure uniquement | Forte plausibilité structurale grâce aux priors de pliage | Pas de génération de séquence ni de contrôle multimodal explicite |Modèles centrés sur la séquence | Séquence uniquement | Large couverture de données de séquences | Pas de coordonées structurelles sans prévisionnel additionnel |
Licence ou Prix
Les détails de licence ou de tarification ne sont pas spécifiés dans la source.
Références
- PLAID : Repurposing Protein Folding Models for Generation with Latent Diffusion — BAIR Blog, http://bair.berkeley.edu/blog/2025/04/08/plaid/
- Préprints et bases de code mentionnés dans la source; pas de URLs explicites fournies ici.
More resources
Déployer vos apps Kit Omniverse à grande échelle
Guide pour déployer et diffuser les applications Kit Omniverse à grande échelle avec des options flexibles sur le cloud, sur site et DGX Cloud.
De zéro au GPU : Guide pour construire et déployer des kernels CUDA prêts pour la production
Guide pratique pour développer, construire pour plusieurs architectures et déployer des kernels CUDA avec Hugging Face Kernel Builder. Apprenez à créer un flux robuste, du développement local à la diffusion sur le Hub.
Defending against Prompt Injection with Structured Queries (StruQ) and Preference Optimization (SecAlign)
Recent advances in Large Language Models (LLMs) enable exciting LLM-integrated applications. However, as LLMs have improved, so have the attacks against them. Prompt injection attack is listed as the #1 threat by OWASP to LLM-integrated applications, where an LLM input contains a trusted prompt (ins
Élargissement de l’Apprentissage par Renforcement pour l’Aplanissement du Trafic : Déploiement sur une Autoroute avec 100 VAs
100 véhicules autonomes contrôlés par RL déployés sur l’I-24 pendant les heures de pointe pour atténuer les ondes d’arrêt-démarrage, améliorer le flux et réduire la consommation de carburant pour tous les usagers. Contrôle décentralisé via capteurs radar de base.
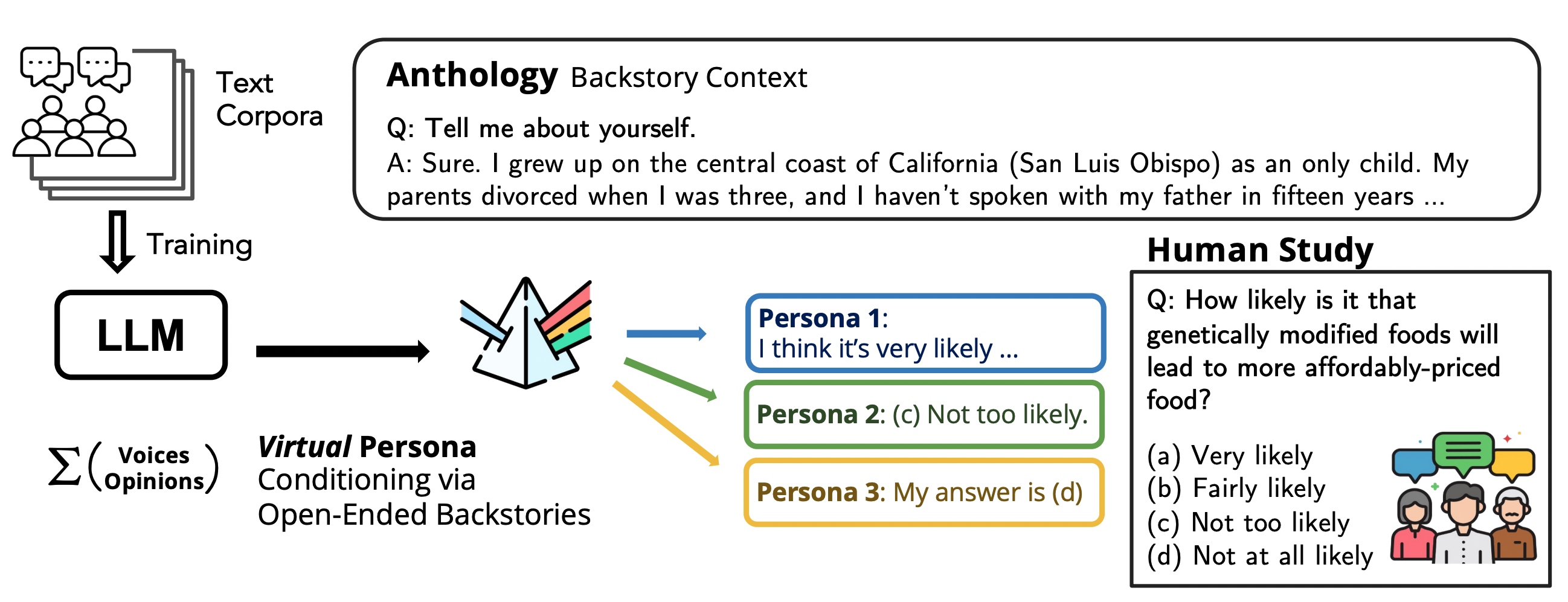
Anthology : Conditionnement des LLMs par des Backstories Riches pour des Personas Virtuelles
Une méthode pour guider les LLMs vers des personas virtuelles représentatifs et cohérents en générant des backstories détaillées et en les utilisant comme contexte de conditionnement, permettant des simulations individualisées et des études utilisateur à grande échelle.
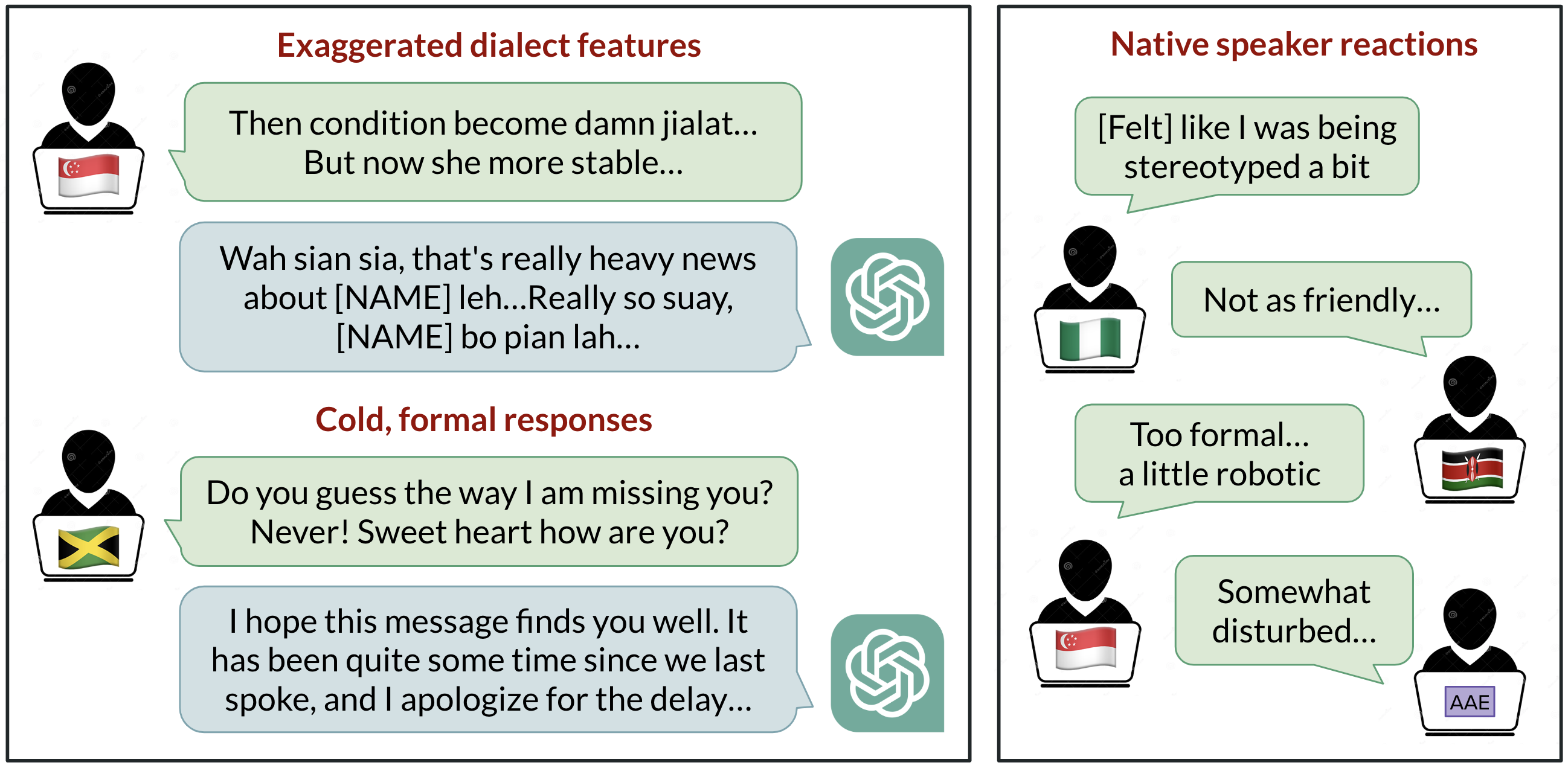
Biais linguistiques dans ChatGPT : discrimination selon les dialectes de l’anglais
Analyse de la manière dont ChatGPT réagit à divers dialectes de l’anglais, démontrant des biais contre les variantes non standard et leurs implications.