Accélérer l’Inference de la Structure des Protéines de Plus de 100x avec le NVIDIA RTX PRO 6000 Blackwell Server Edition
Sources: https://developer.nvidia.com/blog/accelerate-protein-structure-inference-over-100x-with-nvidia-rtx-pro-6000-blackwell-server-edition, https://developer.nvidia.com/blog/accelerate-protein-structure-inference-over-100x-with-nvidia-rtx-pro-6000-blackwell-server-edition/, NVIDIA Dev Blog
TL;DR
- Le NVIDIA RTX PRO 6000 Blackwell Server Edition accélère l’inférence end-to-end de la structure des protéines sur un seul serveur via OpenFold, avec une précision équivalente à AlphaFold2.
- Des benchmarks montrent jusqu’à 138x plus rapide que AlphaFold2 et environ 2,8x plus rapide que ColabFold; des accélérations d’alignement et d’inférence dépassent des centaines de fois les baselines CPU.
- La plateforme offre 96 Go de mémoire à haute bande passante (1,6 To/s), une fonctionnalité MIG qui transforme une carte unique en quatre GPUs et des flux de travail entièrement résidents sur le GPU pour les MSAs et ensembles protéiques.
Contexte et historique
Comprendre la structure des protéines est essentiel pour accélérer la découverte de médicaments, l’ingénierie enzymatique et les biotechnologies agricoles. Depuis AlphaFold2, l’inférence IA pour les structures protéiques a changé la donne, mais des goulets d’étranglement demeurent et peuvent augmenter les coûts et les délais. La génération d’MSA basée sur CPU et une inférence GPU inefficace ralentissent encore les pipelines. Dans ce contexte, NVIDIA et ses laboratoires de biologie numérique ont développé des accélérations qui rendent possible une inference de structure protéique rapide et entièrement sur le GPU avec OpenFold et TensorRT, sans perte d’exactitude par rapport à AlphaFold2. Post du blog NVIDIA. Les charges de travail modernes exigent des MSAs à l’échelle métagénomique, des raffinement itératifs et des calculs d’ensembles qui prennent souvent des heures. Passer ces workloads à l’échelle du protéome sur une infrastructure CPU est généralement prohibitif. L’accélération par GPU démontrée avec le RTX PRO 6000 Blackwell répond à ce problème en déchargeant le flux de travail entier sur un serveur de pointe, permettant des analyses protéiques à grande échelle dans les laboratoires, les plateformes logicielles et les environnements cloud. Des comparaisons montrent que le MMseqs2-GPU peut surpasser les méthodes CPU habituelles d’un ordre de grandeur, illustrant le potentiel de la révolution GPU en bioinformatique.
Ce qui est nouveau
Le RTX PRO 6000 Blackwell Server Edition introduit une combinaison d’innovations matériel et logiciel pour pousser l’inférence de protéines à des vitesses sans précédent :
- Une plateforme GPU de haut niveau pour l’inférence end-to-end avec OpenFold, accélérée par de nouvelles instructions et des optimisations TensorRT.
- Une intégration efficace de MMseqs2-GPU qui accélère fortement la génération de MSAs et le pré-traitement.
- Optimisations TensorRT sur mesure pour OpenFold, produisant des gains de vitesse significatifs par rapport à une configuration OpenFold de base.
- Validation sur des benchmarks standard, dont 20 cibles CASP14, montrant des prédictions avec des TM-scores identiques à ceux d’AlphaFold2 mais avec des temps d’exécution considérablement réduits.
- Mémoire de 96 Go et MIG : permettre des ensembles volumineux et MSAs entièrement sur le GPU; MIG permet à une seule RTX PRO 6000 d’agir comme quatre GPUs virtuels, facilitant le partage des ressources entre plusieurs utilisateurs ou workflows.
- Disponibilité dès maintenant : le RTX PRO 6000 Blackwell Server Edition est disponible via les NVIDIA RTX PRO Servers et les instances cloud des principaux fournisseurs.
Pourquoi c’est important (impact pour les développeurs/entreprises)
Pour les développeurs de plateformes dédiées à la découverte de médicaments, à la protéomique ou à la préparation pour les pandémies, cette avancée apporte des bénéfices réels :
- Des itérations plus rapides permettent de tester des hypothèses et de prendre des décisions plus rapidement.
- Le folding à l’échelle du protéome devient réalisable sur un seul serveur, réduisant le besoin de grands clusters CPU et facilitant l’analyse à grande échelle.
- L’exécution complète sur GPU réduit les transferts de données et accélère les cycles de recherche et développement.
- Le MIG permet une collaboration multi-utilisateur sur un serveur partagé sans perte de débit, optimisant l’utilisation des ressources dans les laboratoires et les environnements cloud.
- La combinaison OpenFold, cuEquivariance, TensorRT et MMseqs2-GPU sur une seule machine fixe une nouvelle référence pour la vitesse d’inférence des structures protéiques et ses implications pour la recherche et les applications industrielles. Selon NVIDIA, ces accélérations maintiennent l’exactitude des prédictions par rapport à AlphaFold2. Post du blog NVIDIA.
Détails techniques ou Mise en œuvre
Les performances reposent sur une combinaison de capacités hardware et d’optimisations software :
- Base matérielle : 96 Go de mémoire à haute bande passante (1,6 To/s) avec MIG, ce qui permet d’exécuter l’intégralité du flux de travail sur le GPU, y compris les MSAs volumineuses et les ensembles protéiques. MIG permet à un RTX PRO 6000 d’être partitionné en quatre GPUs virtuels.
- Stack logiciel et optimisations : les instructions nouvelles et les optimisations TensorRT pour OpenFold, avec MMseqs2-GPU, produisent des gains substantiels. OpenFold bénéficie d’un tuning TensorRT sur mesure qui donne environ 2,3x d’amélioration par rapport à une exécution OpenFold de base.
- Performance démontrée : sur des cibles CASP14, l’inférence sur RTX PRO 6000 Blackwell est 138x plus rapide que AlphaFold2 et environ 2,8x plus rapide que ColabFold, tout en conservant des TM-scores identiques. Dans un benchmark d’alignement, MMseqs2-GPU sur un seul L40S est ~177x plus rapide que JackHMMER sur CPU 128 cœurs, et jusqu’à 720x plus rapide lorsqu’il est réparti sur huit GPUs L40S.
- Intégration du flux de travail : un exemple complet montre comment déployer le OpenFold2 NIM sur une machine locale, générer des requêtes d’inférence et utiliser l’endpoint local pour produire des prédictions. Cela rend possible le folding sur un seul serveur avec une vitesse de classe mondiale.
- Disponibilité et déploiement : le RTX PRO 6000 Blackwell Server Edition est disponible aujourd’hui via les NVIDIA RTX PRO Servers et les instances cloud des principaux fournisseurs. NVIDIA invite les partenaires à collaborer pour déployer des folding de protéines à vitesse et à échelle inégalées. Les remerciements incluent des chercheurs de NVIDIA, de l’University of Oxford et de Seoul National University.
Points clés à retenir
- L’inférence end-to-end des structures protéiques peut être exécutée sur un seul serveur avec le RTX PRO 6000 Blackwell, offrant une vitesse de classe mondiale sans perte d’exactitude par rapport à AlphaFold2.
- Les gains touchent l’ensemble du pipeline : MMseqs2-GPU accélère la génération de MSAs, TensorRT optimise l’inférence OpenFold et l’ensemble des technologies produit des bénéfices importants par rapport aux flux CPU.
- Les benchmarks montrent jusqu’à 138x plus rapide que AlphaFold2 et environ 2,8x plus rapide que ColabFold, avec des alignements 177x plus rapides sur un seul L40S et jusqu’à 720x lorsque l’on scale sur plusieurs GPUs.
- Des ressources telles que 96 Go de mémoire et MIG permettent des workflows volumineux et résidents sur le GPU, avec partage multi-utilisateur sans perte de performance.
- La disponibilité est immédiate via les serveurs RTX PRO et les fournisseurs cloud, ouvrant la voie à l’adoption dans les laboratoires et les environnements d’entreprise.
FAQ
-
- **Q : Que peut faire le RTX PRO 6000 Blackwell Server Edition ?**
Il permet une inference end-to-end de structures protéiques avec OpenFold sur un seul serveur, en tirant parti de MMseqs2-GPU, TensorRT et d’autres accélérations pour offrir des gains importants par rapport à AlphaFold2 et ColabFold, tout en préservant l’exactitude. - **Q : Quelle est la vitesse par rapport aux baselines ?** **A :** Jusqu’à 138x plus rapide que AlphaFold2 et environ 2,8x plus rapide que ColabFold pour le flux OpenFold; MMseqs2-GPU sur un seul L40S est ~177x plus rapide que JackHMMER sur CPU 128 cœurs, et jusqu’à 720x lorsqu’il est déployé sur huit GPUs. - **Q : Quels éléments matériels soutiennent ces gains ?** **A :** 96 Go de mémoire à haute bande passante (1,6 To/s), MIG pour partitionner un GPU en quatre virtuels, et une pile logicielle incluant cuEquivariance, TensorRT et MMseqs2-GPU. - **Q : Est-ce disponible dès maintenant ?** **A :** Oui. Le RTX PRO 6000 Blackwell Server Edition est disponible aujourd’hui via les NVIDIA RTX PRO Servers et les instances cloud des principaux fournisseurs. - **Q : Où trouver plus d’informations ou lancer une déploiement ?** **A :** Consultez le post du blog NVIDIA pour les benchmarks et les directives, puis contactez les partenaires NVIDIA pour planifier une mise en œuvre adaptée à votre laboratoire ou votre entreprise. [Post du blog NVIDIA](https://developer.nvidia.com/blog/accelerate-protein-structure-inference-over-100x-with-nvidia-rtx-pro-6000-blackwell-server-edition/).
Références
More news
NVIDIA HGX B200 réduit l’intensité des émissions de carbone incorporé
Le HGX B200 de NVIDIA abaisse l’intensité des émissions de carbone incorporé de 24% par rapport au HGX H100, tout en offrant de meilleures performances IA et une efficacité énergétique accrue. Cet article résume les données PCF et les nouveautés matérielles.
Scaleway rejoint les Fournisseurs d’Inference de Hugging Face pour une Inférence Serverless et Faible Latence
Scaleway est désormais un Fournisseur d’Inference pris en charge sur Hugging Face Hub, permettant l’inférence serverless directement sur les pages de modèles avec les SDK JS et Python. Accédez à des modèles open-weight populaires et bénéficiez d’une latence réduite.
Prévoir les phénomènes météorologiques extrêmes en quelques minutes sans superordinateur : Huge Ensembles (HENS)
NVIDIA et le Lawrence Berkeley National Laboratory présentent Huge Ensembles (HENS), un outil IA open source qui prévoit des événements météorologiques rares et à fort impact sur 27 000 années de données, avec des options open source ou prêtes à l’emploi.
Comment réduire les goulots d’étranglement KV Cache avec NVIDIA Dynamo
NVIDIA Dynamo déporte le KV Cache depuis la mémoire GPU vers un stockage économique, permettant des contextes plus longs, une meilleure concurrence et des coûts d’inférence réduits pour les grands modèles et les charges AI génératives.
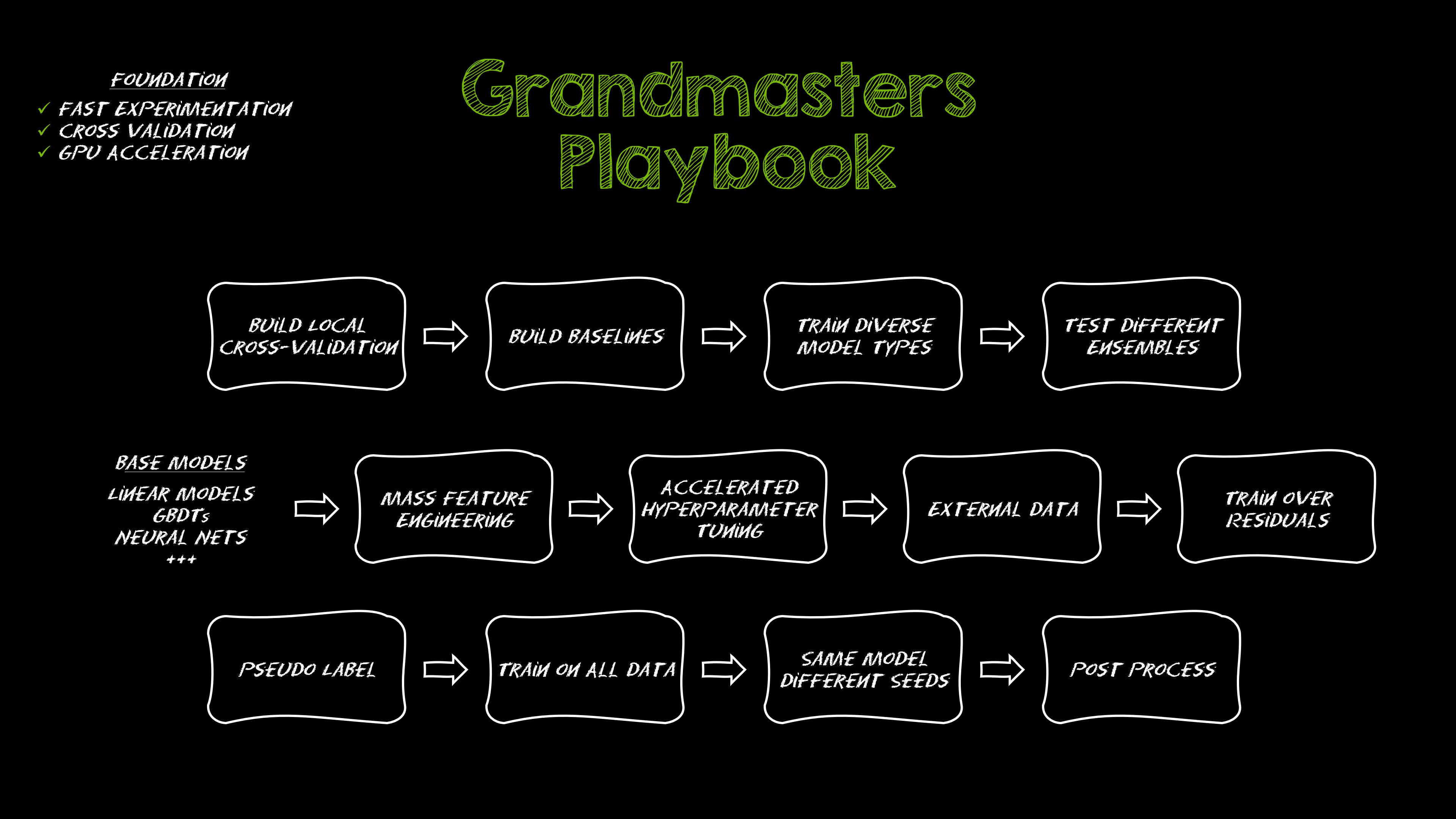
Le Playbook des Grands Maîtres Kaggle: 7 Techniques de Modélisation pour Données Tabulaires
Analyse approfondie de sept techniques éprouvées par les Grands Maîtres Kaggle pour résoudre rapidement des ensembles de données tabulaires à l’aide d’une accélération GPU, des baselines divers à l’assemblage et à la pseudo-étiquetage.
Microsoft transforme le site Foxconn en data center Fairwater AI, présenté comme le plus puissant au monde
Microsoft dévoile des plans pour un data center Fairwater AI de 1,2 million de mètres carrés au Wisconsin, abritant des centaines de milliers de GPU Nvidia GB200. Le projet de 3,3 milliards de dollars promet un entraînement IA sans précédent.