PLAID : Réutiliser les modèles de pliage des protéines pour une génération multimodale par diffusion latente
Sources: http://bair.berkeley.edu/blog/2025/04/08/plaid, bair.berkeley.edu
TL;DR
- PLAID est un modèle génératif multimodal qui produit conjointement la séquence 1D des protéines et leur structure 3D en échantillonnant l’espace latent d’un modèle de pliage, via diffusion.
- Il s’entraîne sur des données de séquences uniquement, utilisant un processus de diffusion sur les embeddings latents pour générer séquence et structure lors de l’inférence.
- Des invites peuvent spécifier la fonction et l’organisme, permettant un contrôle compositionnel du design par interface textuelle.
- L’approche emploie ESMFold comme backbone gelé et introduit CHEAP pour compresser les embeddings conjoints, gérant la taille et la régularisation des espaces latents.
- PLAID suggère un cadre plus large pour la génération multimodale, où une modalité plus abondante peut guider une modalité moins abondante, ouvrant des perspectives de conception en biologie et au-delà. Pour plus de détails, consultez le billet BAIR Berkeley sur PLAID : PLAID sur BAIR Berkeley.
Contexte et arrière-plan
Le prix Nobel 2024 attribué à AlphaFold2 a mis en lumière le rôle crucial de l’IA en biologie, marquant une étape majeure dans la science des protéines et la prédiction structurelle. Dans ce cadre, PLAID propose une nouvelle direction : au lieu de générer des protéines dans une seule modalité, il s’agit d’apprendre et d’échantillonner l’espace latent d’un modèle de pliage pour produire des paires séquence/structure cohérentes. L’idée centrale est d’utiliser un modèle de diffusion qui opère dans l’espace latent d’un modèle de pliage des protéines, afin que la génération soit guidée par la séquence et les coordonnées structurelles pendant l’inférence. Une observation clé est l’asymétrie entre la disponibilité des données pour les séquences et pour les structures. Les bases de données de séquences sont bien plus vastes que celles des structures, car les séquences sont moins coûteuses à obtenir expérimentalement. PLAID exploite cette disparité en entraînant un générateur qui n’a besoin que de séquences pour apprendre l’espace d embedding et décode ensuite la structure lors de l’inférence, en utilisant les poids d’un modèle de pliage pré-entraîné et gelé. PLAID s’appuie sur ESMFold, un successeur d’AlphaFold2 qui remplace une étape de récupération par un modèle de langage protéique. Pendant l’entraînement, seul le contenu en séquences est nécessaire pour obtenir les embeddings; à l’inférence, la structure est décodée à partir de l’embedding échantillonné. Les auteurs insistent sur le fait que ce déploiement permet d’utiliser les connaissances structurelles contenues dans les poids du modèle de pliage pré-entraîné pour les tâches de conception protéique. PLAID introduit également CHEAP (Compressed Hourglass Embedding Adaptations of Proteins), un mécanisme de compression pour l embedding joint séquence–structure. L’objectif est de gérer des espaces latents très grands et fortement activés associés aux modèles transformeurs, tout en préservant la fidélité de génération.
Ce qui est nouveau
- Génération multimodale de co-conception : PLAID aborde directement la génération conjointe de séquence discrète et de coordonnées structurelles continues.
- Diffusion latente sur l’espace de pliage : échantillonnage de l’espace latent d’un modèle de pliage, puis décodage de la structure via des poids de pliage gelés.
- Entraînement sur les séquences uniquement : l’apprentissage utilise uniquement des données de séquence pour obtenir les embeddings, la structure étant générée à l’inférence.
- Invite fonction et organisme : les invites textuelles guident la génération selon la fonction et l’organisme souhaités.
- CHEAP pour la compression : solution pratique pour gérer les grands espaces latents et les activations élevées des modèles de transformeurs.
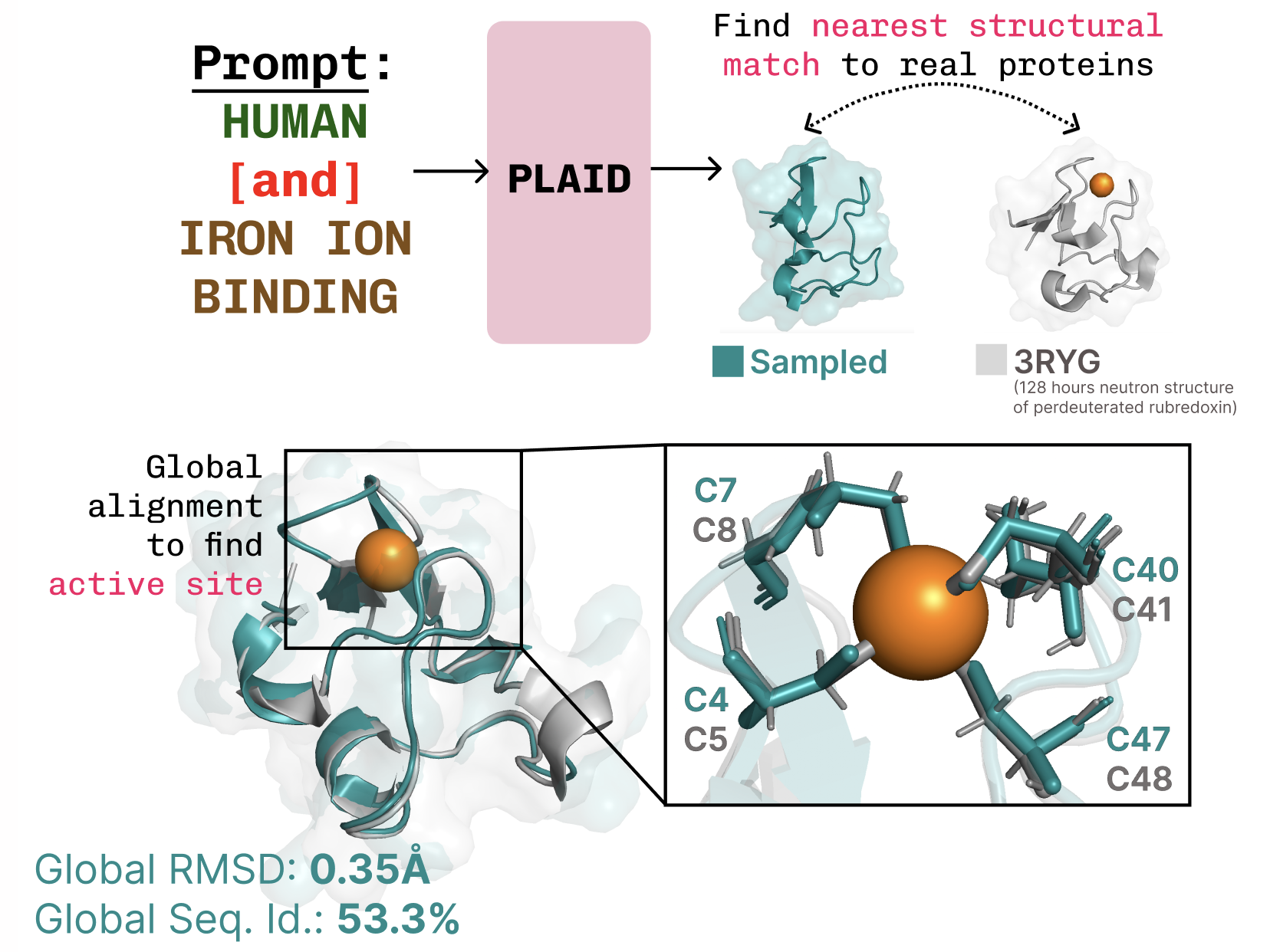
- Motifs protéiques métalliques et membranes : le modèle apprend des motifs de coordination typiques des métallo-protéines (ex. coordination cystéine-Fe), tout en préservant une grande diversité de séquences et en générant des protéines membranaires avec un cœur hydrophobe.
- Perspective multimodale : bien que démontré pour la séquence–structure, la méthode peut être adaptée à d’autres modalités où une prédiction relie une modalité plus abondante à une moins abondante.
Pourquoi cela compte (impact pour les développeurs/entreprises)
- Contrôle accru via des invites : permettre des invites par fonction et organisme facilite un flux de conception plus contrôlé, réduisant le nombre d’essais et d’erreurs lors de la phase initiale et orientant la génération vers des classes fonctionnelles et des hôtes organiques souhaités.
- Efficacité des données via diffusion latente : l’entraînement sur de vastes bases de données de séquences est réalisable et scalable, tirant parti d’un volume important de données.
- Voie pratique vers une conception multimodale : pouvoir générer à la fois la séquence et la structure à partir d’un même embedding latent suggère une approche modulaire pour les futures outils d’IA en biologie.
- Potentiel pour des systèmes plus complexes : avec les avancées dans les prédictions de structures en complexes, la base multimodale PLAID pourrait être étendue à des tâches de conception plus complexes.
Détails techniques ou Mise en œuvre
- Diffusion latente dans l’espace de pliage : PLAID apprend à échantillonner l’espace latent d’un modèle de pliage et utilise ces échantillons pour générer des protéines valides, la structure étant décodée à partir des embeddings via les poids gelés.
- Décodage avec poids fixes : à l’inférence, le modèle de pliage sert de décodeur pour traduire les échantillons latents en structures atomiques complètes.
- Backbone ESMFold : l’approche s’appuie sur ESMFold comme fondation du pliage, tirant parti d’un modèle de langage protéique pour préparer des priors structurels.
- Entraînement sur les séquences : le régime d’entraînement nécessite uniquement des données de séquence pour obtenir les embeddings, profitant de la disponibilité massive.
- CHEAP : mécanisme de compression pour les embeddings conjoints séquence–structure, facilitant l’apprentissage et l’échantillonnage tout en conservant la puissance representational.
- Activations massives et compressibilité : l’étude du espace latent révèle des canaux avec des activations très élevées à travers de nombreuses couches, mais l’espace latent est globalement très compressible, ce qui facilite la génération efficace.
- Généralisation multimodale : le cadre peut être adapté à d’autres modalités où une prédiction peut relier une modalité abondante à une moins abondante.
Conclusions et points clés
- PLAID montre une voie viable pour le design protéique multimodal en combinant diffusion latente et décodage par un modèle de pliage gelé.
- L’entraînement sur les séquences seules peut produire des paires séquence–structure cohérentes, tirant parti des vastes bases de données de séquences.
- Les invites par fonction et organisme offrent une interface pratique pour orienter le design, avec un potentiel futur pour un contrôle entièrement textuel.
- CHEAP apporte une solution pratique pour gérer les grands espaces latents et les activations élevées associées aux transformeurs.
- Le travail s’inscrit dans une tendance plus large où les priors des grands modèles fournissent perception, raisonnement et génération pour les tâches en aval.
FAQ
-
- **Q : En quoi PLAID se distingue-t-il des générateurs protéiques traditionnels ?**
PLAID génère à la fois la séquence et la structure en échantillonnant l’espace latent d’un modèle de pliage et en décodant la structure avec des poids gelés; il s’entraîne sur des séquences uniquement et accepte des invites par fonction et organisme. - **Q : Quels données faut-il pour entraîner PLAID ?** **A :** Seulement des données de séquence pour obtenir les embeddings, en profitant de l’immense base de données de séquences disponible. - **Q : Quel est le rôle des invites dans PLAID ?** **A :** Les invites guident la génération en fonction de la fonction et de l’organisme ciblés, orientant le design. - **Q : Qu’est-ce que CHEAP et pourquoi est-ce utilisé ?** **A :** CHEAP est une compression des embeddings conjoints de séquence et structure; aide à gérer les espaces latents vastes et les activations élevées, facilitant l’apprentissage et la génération.
Références
- PLAID et CHEAP décrits par BAIR Berkeley : http://bair.berkeley.edu/blog/2025/04/08/plaid
More news
Prompting pour la précision avec Stability AI Image Services sur Amazon Bedrock
Bedrock intègre Stability AI Image Services avec neuf outils pour créer et modifier des images avec précision. Apprenez les techniques de prompting adaptées à l’entreprise.
Évoluer la production visuelle avec Stability AI Image Services dans Amazon Bedrock
Stability AI Image Services est désormais disponible dans Amazon Bedrock, offrant des capacités d’édition d’images prêtes à l’emploi via l’API Bedrock et étendant les modèles Stable Diffusion 3.5 et Stable Image Core/Ultra déjà présents.
Mercury, modèles de base d'Inception Labs disponibles sur Amazon Bedrock Marketplace et SageMaker JumpStart
AWS annonce Mercury et Mercury Coder, les modèles de base d'Inception Labs, accessibles via Amazon Bedrock Marketplace et SageMaker JumpStart, offrant une génération par diffusion ultra-rapide pour les charges IA génératives sur AWS.
Déployer des applications Omniverse Kit à grande échelle avec Kit App Streaming
Découvrez comment Kit App Streaming réduit les frictions d’installation et permet le streaming navigateur d’applications Kit basées sur OpenUSD via des GPUs RTX, avec des options de déploiement flexibles on‑premise et cloud.
Poids Ouverts et IA pour tous : Lancement des modèles à poids ouverts pour élargir l’accès
OpenAI met l’accent sur une mission visant à mettre l’IA entre les mains du plus grand nombre et lance aujourd’hui les modèles à poids ouverts les plus performants — une étape majeure pour rendre l’IA avancée plus ouverte, flexible et accessible.
Défense contre l’injection de prompts avec StruQ et SecAlign : requêtes structurées et optimisation des préférences
BAIR de Berkeley présente StruQ et SecAlign comme défenses contre l’injection de prompts dans les applications LLM, avec Front-End Sécurisé, stratégies d’entraînement et améliorations de robustesse.